El problema del maldito RAM y las bases de datos enormes
No se si es el caso de todos, pero por mi parte, cada vez que llego a un nuevo trabajo, me pasan el peor computador de la oficina, usualmente 4 Gb de RAM a lo sumo (y con Windows de 32 bits si tienes muy mala suerte, como me pasó a mi una vez). Como ocupo R para todo, se me hace muy difícil trabajar con bases de datos grandes, a veces mayores a la memoria asignada a R. Este tutorial sirve también si tenemos una base de datos enorme
A continuación vamos a revisar cuanta memoria RAM tiene asignada R en el sistema, y a crear una base de datos SQLite de la cual vamos a poder extraer datos y realizar análisis que pueden ser manejados en nuestra memoria RAM, todo con la ayuda de una función que adapté de este gist de vnijs
En este tutorial vamos a usar el formato de base de datos SQLite por que es fácil de configurar y usar, y puede ser implementado localmente en simples pasos. Otros formatos posibles serian PostgreSQL y MySQL, pero sería un poco más complicado
Para poder usar este tutorial se requiere de un conocimiento intermedio-avanzado de R, y entender un poco el mundo del SQL
Primero lo primero: ¿Cuanta memoria tengo disponible?
Para ver cuanta memoria tiene R disponible vamos a usar el comando memory.size()
memory.limit()## [1] 16269Como podemos ver, tengo un total de 16 Gb de memoria disponible (aprox. 16000 Mb), que es lo que tiene el computador que estoy utilizando.
Nota: Si estas ocupando un sistema operativo de 32 bits, tu limite de memoria no podrá exceder los 2-3 Gb, no importa cuanto RAM tengas instalado.
CSV o TXT a SQLlite
Ahora supongamos que tenemos una base de datos en un csv o txt ENORME. Para efectos de este tutorial voy a ocupar la base de datos del censo de población y vivienda de Chile (año 2017), no es una base de datos tan grande pero para efectos de este tutorial servirá, la pueden descargar de esta pagina web del Instituto Nacional de Estadísticas de Chile.
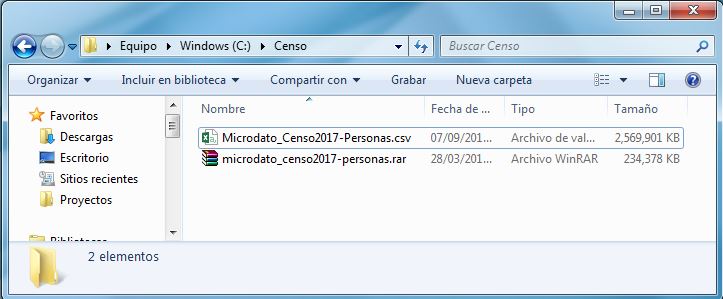
Adentro viene un .rar que descomprimiremos para obtener la base de datos en .csv, como podemos ver, pesa aproximadamente 2.5 Gb, no es tanto, pero si estuviéramos ocupando un computador realmente malo no podríamos trabajarla en R, y abrirla en excel puede ser algo difícil.
La base de datos la voy a descomprimir en una carpeta llamada “Censo” en el disco C:/
Los benditos paquetes
Asegúrate de instalar estos 5 paquetes:
- readr: Para leer el archivo delimitado por comas (csv) por tabulaciones o por el limite que sea
- DBI, RSQLite: Para comunicarnos con las bases de datos y usar SQLite
- dplyr y dbplyr: Para sacar información de la base de datos
library(readr)
library(DBI)
library(RSQLite)
library(dplyr)
library(dbplyr)¿Que tan grande es mi base de datos? ¿Cuales son sus características?
Primero definimos la ruta de nuestra base de datos, le vamos a poner delim_file:
delim_file <- "C:/Censo/Microdato_Censo2017-Personas.csv"Para ver sus características vamos a leer sus primeras 2 lineas, para eso vamos a usar la función readLines
readLines(delim_file, n=2)## [1] "REGION;PROVINCIA;COMUNA;DC;AREA;ZC_LOC;ID_ZONA_LOC;NVIV;NHOGAR;PERSONAN;P07;P08;P09;P10;P10COMUNA;P10PAIS;P11;P11COMUNA;P11PAIS;P12;P12COMUNA;P12PAIS;P12A_LLEGADA;P12A_TRAMO;P13;P14;P15;P15A;P16;P16A;P16A_OTRO;P17;P18;P19;P20;P21M;P21A;P10PAIS_GRUPO;P11PAIS_GRUPO;P12PAIS_GRUPO;ESCOLARIDAD;P16A_GRUPO;REGION_15R;PROVINCIA_15R;COMUNA_15R;P10COMUNA_15R;P11COMUNA_15R;P12COMUNA_15R"
## [2] "15;152;15202;1;2;6;13225;1;1;1;1;1;73;1;98;998;3;15101;998;1;98;998;9998;98;2;4;6;2;1;2;98;7;98;98;98;98;9998;998;998;998;4;2;15;152;15202;98;15101;98"Ya con esta información podemos ver los nombres de los encabezados y ver que el separador (delim) es un punto y coma (;)
Para obtener el número de filas de una base de datos grande R no nos va a servir, ya que no la vamos a poder cargar, así que utilizaremos un comando de Powershell (get content) en el caso de estar en Windows o un comando de sistema (wc) en el caso de estar en Linux.
Para saber cuantas filas tiene mi archivo (esto se demora un poco):
En Windows:
total_records <- system2("powershell", args = c("Get-content", delim_file, "|", "Measure-Object", "–Line"),
stdout = TRUE)
total_records <- as.numeric(gsub("[^0-9]", "", paste(total_records, collapse = ""))) - 1
total_records## [1] 1757400317574003 registros o lineas (dejando fuera el encabezado), ese el numero de personas que registró el censo del año 2017, así que estamos bien.
Nota: Si tienes Windows 10 no debería haber problemas, pero si tienes Windows 7 u 8 te recomiendo ver que versión de powershell tienes instalada estas instrucciones de Microsoft e instalar powershell 5.1 siguiendo este link tambien de Microsoft
En Linux sería algo así:
total_records <- system2("wc", args = c("-l", delim_file), stdout = TRUE) %>%
sub(normalizePath(delim_file), "", .) %>%
as.integer() %>%
{
. - 1
}Crear la ruta a la base de datos
Para crear la base de datos vamos a generar una ruta a un futuro archivo de extensión .sqlite3,
Definimos la ruta y el nombre de la tabla dentro de la base de datos, necesitaremos este nombre para extraer datos en el futuro, otra gracia de estas bases de datos es que pueden contener varias tablas dentro de ellas.
table_name_censo <- "censo2017"
sqlite_file_censo <- "C:/Censo/censo2017.sqlite3"La prometida funcion
Teniendo el numero total de filas del archivo, las rutas definidas y el delimitador (coma, punto y coma, espacio etc), podremos usar la siguiente función:
read_delim2sqlite <- function(delim_file, delim, sqlite_file, table_name, batch_size = 10000, OS = "Windows") {
## establish a connection to the database
condb <- dbConnect(SQLite(), sqlite_file)
## get the total number of records in the file
# in Unix
if (OS == "Unix") {
total_records <- system2("wc", args = c("-l", delim_file), stdout = TRUE) %>%
sub(normalizePath(delim_file), "", .) %>%
as.integer() %>%
{
. - 1
}
} else {
# In windows
total_records <- system2("powershell", args = c("Get-content", delim_file, "|", "Measure-Object", "–Line"), stdout = TRUE)
total_records <- as.numeric(gsub("[^0-9]", "", paste(total_records, collapse = ""))) - 1
}
message("Total records: ", total_records)
## find the number of passes needed based on size of each batch
passes <- total_records %/% batch_size
remaining <- total_records %% batch_size
message("Total Passes to complete: ", passes)
## first pass determines header and column types
dat <- read_delim(delim_file, delim, n_max = batch_size, progress = FALSE) %>% as.data.frame()
if (nrow(problems(dat)) > 0) print(problems(dat))
col_names <- colnames(dat)
col_types <- c(character = "c", numeric = "d", integer = "i", logical = "l", Date = "c") %>%
.[sapply(dat, class)] %>%
paste0(collapse = "")
## write to database table
dbWriteTable(condb, table_name, dat, overwrite = TRUE)
## multiple passes
for (p in 2:passes) {
message("Pass number: ", p, ", Progress:", round(p / passes, 2) * 100, "%")
read_delim(delim_file, delim,
col_names = col_names, col_types = col_types,
skip = (p - 1) * batch_size + 1, n_max = batch_size, progress = FALSE
) %>%
as.data.frame() %>%
dbWriteTable(condb, table_name, ., append = TRUE)
}
if (remaining) {
read_delim(delim_file, delim,
col_names = col_names, col_types = col_types,
skip = p * batch_size + 1, n_max = remaining, progress = FALSE
) %>%
as.data.frame() %>%
dbWriteTable(condb, table_name, ., append = TRUE)
}
## close the database connection
dbDisconnect(condb)
}Se ve algo intimidante la función, pero la verdad no es tan compleja, lo que hace es leer el archivo por partes de cierto tamaño (batch_size), utilizando la función read_delim del paquete “readr” para leer rápidamente el archivo, luego inserta la parte leída en la base de datos SQLite, y repite el proceso hasta la ultima linea del archivo.
Los argumentos de la función son los siguientes:
delim file: Ruta al archivo delimitado (en este caso la BD del censo). delim: Caracter delimitador, en este caso un punto y coma. sqlite_file: Ruta a la base de datos de SQLite, debe terminar con “.sqlite3”. table_name: Nombre de la tabla dentro de la base de datos de SQLite. batch_size: Numero de registros que va a leer en cada iteración. OS: Sistema operativo que estamos utilizando, puede ser “Windows” o “Unix” (Solo probado en Linux, no en Mac).
Mover la csv o txt a la base de datos SQLlite
Con lo que ya tenemos preparado podemos usar la función, en este caso, para asegurarme de no quedarme sin RAM, voy a pasar la base de datos en conjuntos de un millon de lineas (batch_size), por lo que debería terminarse en 18 iteraciones.
Advertencia, esto tardará unos cuantos minutos, vayan a cebarse un mate
read_delim2sqlite(delim_file, delim=";", sqlite_file = sqlite_file_censo,
table_name = table_name_censo, batch_size=1000000)## Total records: 17574003## Total Passes to complete: 17## Parsed with column specification:
## cols(
## .default = col_integer(),
## P18 = col_character()
## )## See spec(...) for full column specifications.## Pass number: 2, Progress:12%## Pass number: 3, Progress:18%## Pass number: 4, Progress:24%## Pass number: 5, Progress:29%## Pass number: 6, Progress:35%## Pass number: 7, Progress:41%## Pass number: 8, Progress:47%## Pass number: 9, Progress:53%## Pass number: 10, Progress:59%## Pass number: 11, Progress:65%## Pass number: 12, Progress:71%## Pass number: 13, Progress:76%## Pass number: 14, Progress:82%## Pass number: 15, Progress:88%## Pass number: 16, Progress:94%## Pass number: 17, Progress:100%Este proceso tan demoroso solo lo haremos una vez. Como podemos ver se crea un archivo .sqlite3, que incluso es un poco más pequeño que nuestro .csv
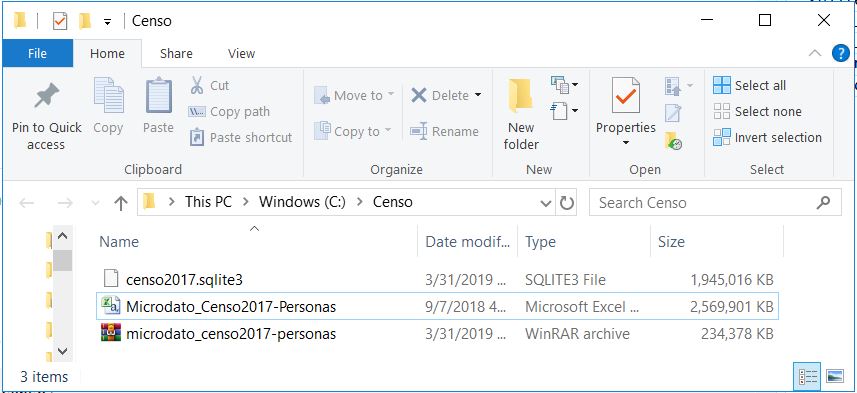
Archivo SQLite creado
Analizar y recoger datos con dplyr
Para empezar a colectar datos de la tabla en primer lugar tengo que hacer una conexión con la base de datos
con <- dbConnect(SQLite(), sqlite_file_censo)
con## <SQLiteConnection>
## Path: C:\Censo\censo2017.sqlite3
## Extensions: TRUEPodemos ver las características de la conexión y el nombre que le asignamos a la base de datos
dbListTables(con)## [1] "censo2017"Creamos un objeto que va a representar a los datos, este objeto no está en nuestro RAM, es solo una conexión, por eso no salen las filas de la base de datos.
datos_db <- tbl(con, "censo2017")
datos_db## # Source: table<censo2017> [?? x 48]
## # Database: sqlite 3.22.0 [C:\Censo\censo2017.sqlite3]
## REGION PROVINCIA COMUNA DC AREA ZC_LOC ID_ZONA_LOC NVIV NHOGAR
## <int> <int> <int> <int> <int> <int> <int> <int> <int>
## 1 15 152 15202 1 2 6 13225 1 1
## 2 15 152 15202 1 2 6 13225 3 1
## 3 15 152 15202 1 2 6 13225 3 1
## 4 15 152 15202 1 2 6 13225 3 1
## 5 15 152 15202 1 2 6 13225 3 1
## 6 15 152 15202 1 2 6 13225 9 1
## 7 15 152 15202 1 2 6 13225 9 1
## 8 15 152 15202 1 2 6 13225 9 1
## 9 15 152 15202 1 2 6 13225 9 1
## 10 15 152 15202 1 2 6 13225 10 1
## # ... with more rows, and 39 more variables: PERSONAN <int>, P07 <int>,
## # P08 <int>, P09 <int>, P10 <int>, P10COMUNA <int>, P10PAIS <int>,
## # P11 <int>, P11COMUNA <int>, P11PAIS <int>, P12 <int>, P12COMUNA <int>,
## # P12PAIS <int>, P12A_LLEGADA <int>, P12A_TRAMO <int>, P13 <int>,
## # P14 <int>, P15 <int>, P15A <int>, P16 <int>, P16A <int>,
## # P16A_OTRO <int>, P17 <int>, P18 <chr>, P19 <int>, P20 <int>,
## # P21M <int>, P21A <int>, P10PAIS_GRUPO <int>, P11PAIS_GRUPO <int>,
## # P12PAIS_GRUPO <int>, ESCOLARIDAD <int>, P16A_GRUPO <int>,
## # REGION_15R <int>, PROVINCIA_15R <int>, COMUNA_15R <int>,
## # P10COMUNA_15R <int>, P11COMUNA_15R <int>, P12COMUNA_15R <int>Ahora podemos ocupar los verbos de dplyr!, por ejemplo, digamos que queremos obtener un vector con las edades simples para hacer un histograma o algo así (la edad en la base de datos del censo es el campo P09).
edades <- datos_db %>%
select(P09)
edades## # Source: lazy query [?? x 1]
## # Database: sqlite 3.22.0 [C:\Censo\censo2017.sqlite3]
## P09
## <int>
## 1 73
## 2 78
## 3 78
## 4 52
## 5 44
## 6 39
## 7 35
## 8 13
## 9 12
## 10 65
## # ... with more rowsPareciera que todo estuviera bien, sin embargo, el objeto sigue siendo una “conexión”, lo que hace R es un “lazy loading” (lazy de perezoso o flojo), es decir, solo realiza una parte de la operación para no demorarse mucho.
Lo podemos confirmar en el environment también:
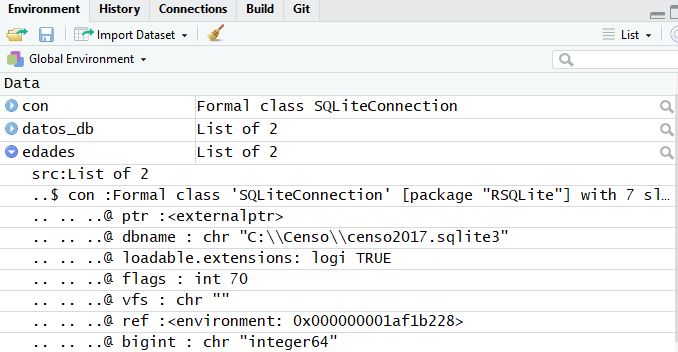
Como podemos ver, el numero de filas ni siquiera esta disponible:
nrow(edades)## [1] NAPara traer esta columna a nuestro RAM necesitamos usar la función collect, recomiendo utilizar esta función siempre, para asegurarnos que estamos trabajando con objetos completos y no con conexiones
edades<-collect(edades)
nrow(edades)## [1] 17574003edades## # A tibble: 17,574,003 x 1
## P09
## <int>
## 1 73
## 2 78
## 3 78
## 4 52
## 5 44
## 6 39
## 7 35
## 8 13
## 9 12
## 10 65
## # ... with 17,573,993 more rowsAhora si!
Otro ejemplo, el campo “P09” se refiere a la edad y el campo “P08” al sexo, supongamos que queremos saber la edad promedio por región de los hombres (Sexo=1). ¡No nos olvidemos del collect!
hombres_edad <- datos_db %>%
filter(P08==1) %>%
group_by(REGION) %>%
summarise(Edad_media = mean(P09))
hombres_edad <- collect(hombres_edad)## Warning: Missing values are always removed in SQL.
## Use `AVG(x, na.rm = TRUE)` to silence this warninghombres_edad## # A tibble: 16 x 2
## REGION Edad_media
## <int> <dbl>
## 1 1 32.1
## 2 2 33.0
## 3 3 33.7
## 4 4 34.6
## 5 5 35.8
## 6 6 35.7
## 7 7 35.9
## 8 8 35.0
## 9 9 35.3
## 10 10 35.1
## 11 11 34.2
## 12 12 35.9
## 13 13 34.2
## 14 14 35.7
## 15 15 33.5
## 16 16 36.8Listo, ya podemos trabajar con gran parte de las base de datos estructuradas, extraer datos y realizar análisis.
¡Ojala les haya gustado el tutorial, no duden en contactarme!


